Na początku sierpnia pisałem o przełomie (jakkolwiek byśmy nie rozumieli tego słowa) w zastosowaniu techniki CRISPR/Cas9 do modyfikowania ludzkich zarodków. Wieść obiła się szerokim echem – w tym szumie łatwo było nie zauważyć innego doniesienia z tego pola badań. 8 sierpnia pismo Nature Biotechnology wycofało mającą zaledwie nieco ponad rok publikację opisującą zastosowanie do redagowania genomu nowego enzymu, tak zwanego NgAgo.
Oryginalna praca była, w momecie publikacji, rewelacją. Aby zrozumieć dlaczego, trzeba rozumieć, w jaki dokładnie sposób działa system CRISPR/Cas9, a także jakie są jego ograniczenia (o tym wszystkim za momencik). Rewelacją jednak przestała być szybko – po początkowym ożywieniu środowiska na ćwierkaczu bardzo szybko pojawiły się próby replikacji wyników. Najpierw nieoficjalne (czyt. nierecenzowane), potem bardziej formalne (na serwerach preprintowych), wreszcie zaś w recenzowanych pismach branżowych.
Piszę „próby”, bo wszystkie grupy próbujące odtworzyć oryginalny wynik pomimo chęci i zapału nie dały rady. Jeden z tych wyników został wreszcie opublikowany przez Nature Biotechnology. Efektem tej pracy był alert redaktorski (ang. expression of concern), który pismo opublikowało pod koniec listopada zeszłego roku. W tym krótkim przypisie redakcja zwracała uwagę czytelników na problemy z powtórzeniem rezultatów pracy. Ostatecznie w sierpniu pismo wycofało pracę za zgodą autorów, cytując jako powód niemożność replikacji.
Przez cały ten okres, od publikacji poprzez wielokrotne próby powtórzenia i odtworzenia doświadczeń, aż po ostateczne wycofanie pracy na początku sierpnia, autorzy oryginału szli w zaparte twierdząc, że badania są solidne, dane rzetelne, a problemy z replikacją są krytyką nie tyle oryginalnych autorów, co osób próbujących iść, nieskutecznie, ich śladem. Autorzy byli też twardo wspierani przez swoją instytucję – pomimo tego, że bardzo wcześnie pojawiły się głosy, że uniwersytet powinien przeprowadzić postępowanie wyjaśniające, czy nie doszło do jakiegoś przekrętu. Postępowania, o ile mi wiadomo, albo nie było albo nie zostało ono ujawnione publicznie.
O bolączkach Cas9
Żeby wyjaśnić, dlaczego NgAgo było przez medialną sekundę takim wielkim halo, wyjaśnić trzeba jak dokładnie działa system CRISPR/Cas9 (czego w detalach nigdy w zasadzie tutaj nie uczyniłem, więc może wreszcie pora). Zacznijmy od podstaw: co w ogóle kryje się pod pojęciem „redagowanie genomu”. Bo łatwo może wpaść w pułapkę postrzegania CRISPR/Cas9 (ale też i innych metod do redagowania genów), jako wszechstronnego narzędzia, które bezbłędnie i skutecznie identyfikuje sekwencję DNA, którą chcemy zmodyfikować, tnie ją, po czym zamienia na taką, jaką chcemy i skleja wszystko z powrotem do kupy. Taki one-stop shop. I gdybyśmy mówili o robocie kuchennym, to może by i tak było. Ale mówimy o systemie biologicznym, który w naturze powstał w nieco innym celu, a który my tylko zaadaptowaliśmy.
W rzeczywistości CRISPR/Cas9 służy jedynie do identyfikacji określonej sekwencji DNA oraz jej przecięcia. Tutaj jego rola się w zasadzie kończy, a do akcji wkraczają różne białka naturalnie obecne w komórce, które stanowią jej system naprawczy. Komórki wykorzystuję dwa główne systemy naprawcze. Jeden jest nieco bardziej efektywny, ale działa po prostu sklejając przecięte nici DNA w sposób, który może być nieco losowy i który nie uwzględnia tego, czy jakiś fragment DNA nie został całkiem usunięty. Skutkiem tego jest najczęściej to, że takie sklejone DNA ma inną sekwencję niż na początku – a jeśli cieliśmy fragment kodujący białko, no to jest duża szansa, że w ten sposób zaburzymy całkiem strukturę tego białka. Tę metodę stosować zatem można do derelegulacji – na przykład jeśli zależy nam tylko na tym, żeby ekspresję jakiegoś białka po prostu wyciszyć (bo jego nadmiar jest patologiczny).
Drugi mechanizm do naprawy DNA wykorzystuje wzorzec sekwencji DNA. W naturze jest to zazwyczej odpowiednik przeciętego genu znajdujący się na siostrzanym allelu. Jednak jeśli do komórki wprowadzimy taki wzorzec wraz z CRISPR/Cas (przez wzorzec mam na myśli długi odcinek nici DNA, który ma sekwencję komplementarną do miejsca cięcia, zwłaszcza do rejonów sąsiedzkich), to ten wzorzec jest wówczas wykorzystywany przez komórkę. Wystarczy jednak, aby gdzieś w środku tego wzorca wprowadzić niewielką zmianę – różnicę kilku nukleotydów – i naprawione DNA będzie wówczas dokładnie takie samo, jak przed modyfikacją (tzn. z działającym białkiem), poza tymi kilkoma podmienionymi nukleotydami. Ten sposób redakcji jest znacznie bardziej precyzyjny, ale też znacznie mniej wydajny (mówimy o różnicy kilku rzedów wielkości).
Jak więc widać, CRISPR/Cas9 jest przede wszystkim maszyną do precyzyjnego cięcia. A nożycami jest w tej parze enzym Cas9. W jaki sposób Cas9 rozpoznaje jednak sekwencję DNA, którą ma pociachać? Służy mu do tego sztuczny odcinek RNA o sekwencji odpowiadającej fragmentowi DNA, który chcemy pociąć. Czyli: Cas9 łapie ten wzorzec RNA (nazywany sgRNA z angielskiego single-guide RNA), a gdy RNA ułoży się wzdłuż nici DNA w miejscu, gdzie sekwencje są komplementarne (a teoretycznie jeśli nie są, to się nie ułoży), wówczas Cas9 przystępuje do dzieła i tnie.
Proste? Nie do końca. Okazuje się bowiem, że Cas9 ma pewne niemałe ograniczenie: tnie sekwencję odpowiadającą sgRNA tylko wtedy, kiedy w DNA jest ona poprzedzona ściśle określoną, krótką sekwencją nukleotydów. Dla najczęściej stosowanego enzymu Cas9, izolowanego z bakterii Streptococcus pyogenes, tzw. SpCas9, ta sekwencja to NGG (gdzie N to dowolny nukleotyd, zaś G to guanina). Cas9 rozpoznaje sekwencje długości 22 nukleotydów, ale jeśli taka sekwencja nie jest poprzedzona przez NGG – nie zostanie przecięta.
Biorąc pod uwagę długość sekwencji rozpoznawanej przez enzym (22 nukleotydy), wymóg obecności tej dodatkowej ściśle określonej sekwencji (tzw PAM, z ang. protospacer adjacent motif) jest sporym ograniczeniem. Co prawda PAM różni się pomiędzy enzymami Cas9 izolowanymi z różnych bakterii – może mieć od 2 do 6 nukleotydów i bardzo różne sekwencje – ale w idealnym świecie chcielibyśmy mieć enzym, który nie ma takiego ograczenia. W obecnej chwili, jeśli chcemu stosować do redakcji genu system CRISPR/Cas9, jesteśmy zmuszeni do dobierania enzymu Cas9 tak, aby jego PAM znajdował się w okolicy cięcia genu (a jeśli chcemy ciąć bardzo precyzyjnie – a zazwyczaj chcemy – to nie mamy specjalnego wyboru, jakie sekwencje będą się wokół tego miejsca znajdować).
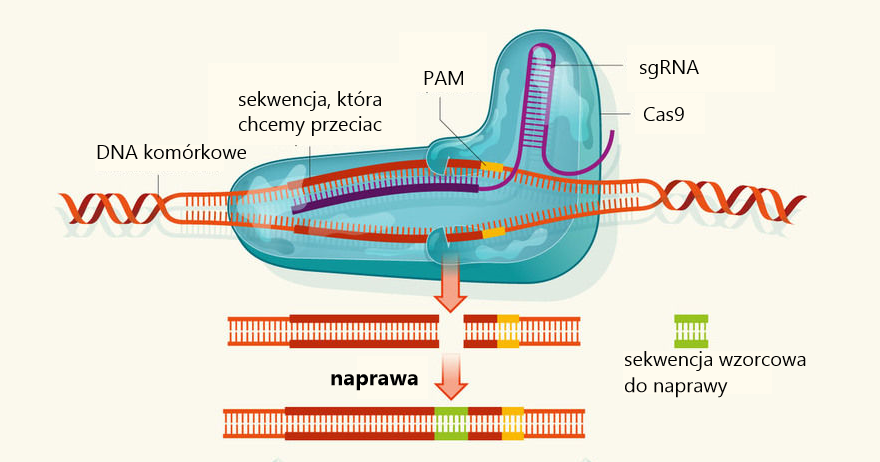
Cas9 ma kilka innych bolączek. Po pierwsze: PAM. Po drugie: długość sekwencji wzorcowej – tylko 22 nukleotydy. Po trzecie: enzym Cas9 dopuszcza nieścisłość w dopasowaniu sgRNA do sekwencji docelowej – to znaczy, że nawet jeśli jeden czy dwa z tych 22 nukleotydów nie pasują, to i tak przetnie (pod warunkiem, że obecna jest sekwencja PAM). Po czwarte: sam proces cięcia jest mało wydajny – wynika to po prostu z kinetyki tego enzymu. Po piąte: Cas9, zwłaszcza najpopularniejszy SpCas9, jest enzymem dość dużym. To oznacza, że nie jest łatwo dostarczyć go do komórki. Większość metod wykorzystuje do tego celu plazmidy – kołowe cząsteczki kwasu nukleinowego, na których zakodowany jest enzym Cas9, a także sgRNA. Taki plazmid wstrzykiwany jest do komórek, które potem same na jego podstawie produkują zarówno Cas9, jak i sgRNA. Jednak rozmiar plazmidów jest ograczniony – SpCas9, które zbudowane jest z ponad 1300 aminokwasów, ma zaś bardzo długą sekwencję własnego DNA, która zajmuje cenne miejsce w plazmidzie.
I tu wreszcie dotarliśmy do sedna – czemu NgAgo, które przecież jest tylko kolejnym bakteryjnym enzymem restrykcyjnym, zostało ochrzczone objawieniem, potężnym rywalem dla Cas9 i, jak mawiają Anglicy, największą rewelacją od czasu wynalezienia chleba tostowego.
O zaletach NgAgo
Ograniczenia Cas9 nie wzięły się z sufitu. Były badaczom znane od początku – i dlatego wielu naukowców pracujących nad rozwojem technologii do redagowania genomu bardzo wcześnie rozpoczęło poszukiwania alternatyw. Do tych zalicza się na przykład izolowanie enzymu Cas9 z innych bakterii niż Streptococcus pyogenes – homologiczne enzymy działają według tej samej zasady, ale mogą mieć inny PAM, być mniejsze, i tak dalej. Inną opcją jest bioinżynieria – laboratoryjne modyfikowanie SpCas9 tak, aby rozszerzyć repertuar dostępnych wersji, które można potem dobierać w zależności od potrzeb.
Alternatywa trudniejsza, ale też bardziej radykalna, to dalsze poszukiwania innych enzymów, która mają zdolność do cięcia DNA i być może potencjał, aby robić to w sposób programowalny. I to właśnie uczynił Chunyu Han z Uniwersytetu w Hebei wraz ze współpracownikami. Han zaczął przeczesywać literaturę fachową w poszukiwaniu wzmianek o innych enzymach pełniących funkcję molekularnych nożyczek. I znalazł – grupę białek z rodziny Argonaute (Ago), które regulują metabolizm kwasów nukleinowych i w organizmach żywych, podobnie do Cas9, są uwikłane w systemy broniące komórki przed obcym DNA.
Han przetestował białka Ago wyizolowane z kilku różnych mikroorganizmów. Białka pochodzące z bakterii Thermus thermophilus (TtAgo) oraz archeonta Pyrococcus furiosus (PfAgo) działały jednak jedynie w temperaturze powyżej 65 stopni Celsjusza – znacznie wyższej niż fizjologiczna. Na żyłę złota badacze trafili, gdy przetestowali Ago pochodzące z archeonta Natronobacterium gregoryi, tzw. NgAgo.
W pracy w Nature Biotechnology Han ze współpracownikami pokazał, że NgAgo ma aktywność enzymatyczną endonukleazy – czyli, że tnie DNA – a także że wykorzystuje do tego jako wzorzec jednoniciowe DNA (a nie RNA, jak Cas9). To jest o tyle ważne, że jednoniciowe DNA w komórkach ssaczych są niezmiernie rzadkie, mniejsze jest więc zagrożenie, że enzym jako wzorzec użyje losową cząsteczkę szwendającą się po komórce. NgAgo ma też wiele innych zalet. Wzorcowa (rozpoznawana) sekwencja ma ok. 24 nukleotydy, co zwiększa szansę na poprawne i precyzyjne rozpoznanie miejsca cięcia. NgAgo nie toleruje nieścisłości w dopasowaniu wzorca do sekwencji, którą chcemy ciąć, dzięki czemu jest znacznie bardziej specyficznym enzymem niż Cas9. Nie wymaga też obecności PAM, co czyni go znacznie bardziej wszechstronnym. Jest prawie o połowę mniejszy – więc łatwiej go dostarczyć do komórki. Wyniki te, chociaż otrzymane w komórkach ludzkich, zostały zademonstrowane tylko in vitro. Ale potencjał wydawał się być ogromny.
NgAgo rozmontowane
Biorąc pod uwagę te wszystkie potencjalne zalety NgAgo, a także to jak wielkim przełomem ten system wydawał się w porównaniu z niekiepskim przecież CRISPR/Cas9, nie powinno być zaskoczeniem, że wiele laboratoriów rzuciło się od razu testować nowy enzym.
Jednym z pierwszych badaczy testujących NgAgo był Dr Gaetan Burgio z Australijskiego Uniwersytetu Narodowego, genetyk pracujący głównie nad chorobami zaraźnymi, który często i gęsto korzysta z technik modyfikowania genetycznego organizmów. Burgio nie próbował zreplikować pracy Hana – zamiast tego zaadaptował protokół laboratoryjny zoptymalizowany we własnym laboratorium tak, aby można było go wykorzystać z nowym enzymem do modyfikacji myszy.
Wyniki tych pierwszych doświadczeń Burgio opisał (wstępnie) na blogu w portalu Medium pod koniec lipca 2016 roku – zaledwie półtora miesiąca po publikacji Hana. Rezultaty były raczej rozczarowujące: wyniki wskazywały na to, że enzym działa w temperaturze znacznie wyższej niż zaproponowana przez Hana (50 cs 37 stopni Celsjusza). Nie do końca było też jasne, że w ogóle ma aktywność endonukleazową (czyli że tnie DNA). Wyglądało za to na to, że ma aktywność ligazową – czyli że gdy DNA jest już pocięte i komórka zaczyna je naprawiać, NgAgo może do wciąż pociętych końcówek DNA dokleić dodatkowe odcinki.
Burgio w swoim raporcie wspomina, że w trakcie, gdy jego grupa testowała NgAgo, inni naukowcy próbowali powtórzyć oryginalną pracę. Inni testowali enzym w danio pręgowanym – laboratoryjnym rybim modelu. Nikomu nie udało się jednak pokazać, że NgAgo rzeczywiście działa. Na początku sierpnia 2016 Pooran Dewari z Edynburga opisał wyniki ankiety przeprowadzonej wśród ponad 150 badaczy próbujących rozpracować NaAgo. Ankieta nie była już nawet rozczarowaniem – była po prostu klęską.
W ciągu mniej niż dwóch miesięcy historia NgAgo z triumfu zamieniła się w kontrolę strat. Nature News doniosło, że w tym samym czasie gdy Burgio publikował w Medium swoje wstępne wyniki, badacz w hiszpańskim Narodowym Centrum Biotechnologii, Lluis Montoliu, wysłał do swoich kolegów i koleżanek maila apelującego o zaprzestrzanie jakichkolwiek projektów z wykorzystaniem NgAgo, aby ograniczyć straty w środkach, pieniądzach i robociźnie.
W kolejnych miesiącach światło dziennie ujrzało zaś kilka publikacji po kolei debunkujących NgAgo jako nowy, super-wydajny programowalny enzym restrykcyjny. W połowie listopada Jialing Qi ze współpracownikami opisali swoje płonne starania, aby zastosować NgAgo do redagowania genów z danio pręgowanym. Shawn Burgess ze współpracownikami opublikował z kolei pracę będącą skutkiem wysiłków kilku laboratoriów, w których próbowano po pierwsze dokładnie zreplikować oryginalne badanie, ale także, po drugie, przetestować NgAgo w innych systemach – w innych komórkach ludzkich, w mysich macierzystych komórkach embrionalnych, w mysich embrionach, a także w embrionach rybich (suplement opisujący w szczegółach wszystkie wyniki powala na kolana). Wreszcie też Nature Biotechnology opublikowało kolejną pracę opisującą problemy z replikacją wyników Hana. A wraz z tą ostatnią na stronach pisma pojawił się wspomniany przeze mnie wcześniej alert redaktorski.
Do pewnej niemiłej refleksji nad tempem działania nauki – albo może raczej sposobu w jaki jest komunikowana – zmusza nas jednak to, że pomimo już tych raczej bardzo przekonywujących wczesnych dowodów na niepoprawność oryginalnej pracy, publikacja Hana wycofana została dopiero na początku sierpnia 2017 roku. A nie jest tak, że krytycy NgAgo spoczęli po redaktorskim alercie na laurach. W maju tego roku Baisong Lu opublikował w PLOS ONE kolejną pracę pokazującą, że enzym nie działa tak, jak teoretycznie, według Hana, powinien. Swoje wstępne wyniki w publikacją zamienił też wreszcie Burgio – jego praca ukazała się, także w PLOS ONE, w czerwcu.
Znając jednak realia procesu od drugiej strony podkreślić muszę, że ta krytyczna refleksja nie jest skierowana przeciw redakcji Nature Biotechnology, ani nawet specjalnie przeciwko autorom oryginalnej publikacji – zwłaszcza że pomimo okoliczności wygląda na to, że wyniki nie były rezultatem machloj, ale raczej braku pewnej rzetelności oraz ostrożności w interpretacji danych.
Z drugiej jednak strony to, że autorzy broń złożyli dopiero teraz, pod naporem dowodów, wydaje się być w pewnym stopniu jednak spowodowane czynnikami pozanaukowymi.
Zdradzieckie konflikty interestów
Od samego początku bowiem Han twardo twierdził, że wyniki są solidne, a problemy z replikacją wynikają z braku kwalifikacji badaczy próbujących badanie odtworzyć. Wyraźnie zależało mu też na tym, aby innym udało się jednak badanie zreplikować – stąd na przykład już w lecie zeszłego roku dokładny protokół laboratoryjny eksperymentów, a także projekt zastosowanych plazmidów, znalazły się w publicznym repozytorium Addgene.
Te chęci jednak mogłby być motywowane szybko nabytą sławą. Chińskie media ochrzciły Hana kandydatem do Nagrody Nobla, wybrano go wice-prezydentem Stowarzyszenia Nauki i Techniki na Uniwersytecie w Hebei, a nawet – jak donosi teraz Science – obwołano najpiękniejszym nauczycielem w Hebei (chociaż mam podejrzenie, że tłumaczenie może tracić tu na znaczeniu). Podczas gdy badacze – zarówno spoza jak i z samych Chin – nawoływali jednak do upublicznienia wszystkich danych, a także wzywali instytucję, w której Han pracuje, Uniwersytet w Hebei, do przeprowadzenia wewnętrzego dochodzenia, sama uczelnia ogłosiła właśnie otwarcie fundowanego głównie przez lokalne władze nowego centrum redagowania genomu (z wartego 32 miliony dolarów grantu). Trudno tutaj nie dostrzec podstawowego problemu, jakim jest konflikt interesów uczelni, która powinna być pierwsza w kolejce do audytu danych w tak mocno kontestowanej pracy.
Finansowa motywacja, aby opierać się krytyce i – prawdopodobnym – zabiegom redaktorów usiłujących wycofać publikację, nie zniknęła nawet po publikacji alertu redaktorskiego. W styczniu Nature News doniosło o firme zainteresowanej komercjalizacją NgAgo. Intencje firmy są nie do końca jasne – bardzo możliwe, że w NgAgo firma widzi potencjał w dziedzinie zupełnie innej niż redagowanie genomu. Łatwo jednak dostrzec, jak możliwość zdobycia solidnego inwestora mogła autorom oryginalnej pracy dać do myślenia.
Podejrzewać można zatem – chociaż podkreślam, że na tym etapie jest to już moja czysta spekulacja – że z jednej strony autorzy opierali się retrakcji z przyczyn zarówno reputacyjnych jak i finansowych. Z drugiej strony redaktorzy mogli mieć związane ręce – jeśli bowiem nie ma dowodów na manipulację (a nie pomaga, że takie dowody pochodzić mogą tylko od instytucji albo agencji rządowych zajmujących się badaniem naukowych przekrętów, takich jak amerykańskie ORI) i nie jest jasne, że praca jest naukowo niepoprawna, bo nie da się wskazać dokładnie problemu, wówczas do retrakcji może nie dojść nigdy! Biorąc pod uwagę informacje dostępne w domenie publicznej, mam nawet podejrzenie, że decyzja o retrakcji mogła tutaj nie zapaść – i NgAgo mogło podzielić los arsenowych bakterii opisanych pół dekady temu w Science w pracy, o której wiadomo, że w najlepszym wypadku jest objawem naiwnej niekompetencji, a w najgorszym hochsztaplerskim bublem, a mimo tego nie została nigdy wycofana. Może zatem powinniśmy się cieszyć, że światowe środowisko naukowe zmarnowało jedynie rok środków ludzkich, finansowych i intelektualnych, aby dowieść, że nie wszystko złoto, co się świeci.
Ilustracja w nagłówku to Danio rerio, rybka będąca jednym z czynników, które rzuciły NgAgo na kolana. Zdjęcie z flickr, dzięki uprzejmości Thierry Marysael (CC By NC ND 2.0).


Nie tylko CRISPR/Cas…:
https://www.vox.com/science-and-health/2017/10/9/16435802/nocebo-effect-medicine-placebo
PolubieniePolubienie